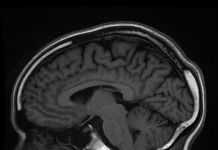
La séquence complète du génome humain des deux chromosomes X et des autosomes de la lignée cellulaire dérivée du tissu féminin a été achevée. Cela inclut les 8 % de la séquence du génome qui manquaient dans le projet original publié en 2001.
Le complet génome humain séquence de l'ensemble des 3.055 milliards de paires de bases a été révélée par le Consortium Telomere-to-Telomer (T2T). Il s'agit de la plus grande amélioration apportée au génome humain de référence publié en 2001 par Celera Genomics et l'International Human Genome Sequencing Consortium. Cette séquence du génome couvrait la plupart des régions euchromatiques tout en laissant de côté les régions d'hétérochromatine ou une représentation erronée. Ces régions représentent 8 % du génome humain enfin révélé. La nouvelle référence T2T-CHM131 comprend une séquence complète pour les 22 autosomes plus le chromosome X. Cette nouvelle séquence de référence a également corrigé de nombreuses erreurs et a ajouté environ 200 millions de pb de nouvelles séquences contenant 2,226 115 copies de gènes, dont XNUMX devraient être des protéines codantes.
Le génome de référence GRCh38.p13 actuel est le résultat de deux mises à jour majeures, l'une en 2013 et l'autre en 2019 sur la séquence du génome de Celera de 2001. Cependant, il avait encore 151 millions de paires de bases de séquences inconnues réparties dans tout le génome, y compris des régions péricentromériques et subtélomériques, des duplications, des puces à ADN ribosomique et de gènes (ADNr), qui sont toutes nécessaires aux processus cellulaires fondamentaux. La nouvelle référence a été nommée T2T-CHM13 car elle provient du séquençage de l'ADN de la lignée cellulaire CHM13 (Complete Hydatiform Mole) et est réalisée par le consortium T2T. La lignée cellulaire est dérivée d'un ovule fécondé anormal ou d'une prolifération de tissu du placenta dans laquelle les femmes semblent être enceintes (fausse grossesse), la séquence ne représente donc que les deux chromosomes X et les autosomes de la femelle. Des technologies de séquences multiples ont été utilisées, telles que les séquenceurs PacBio, Oxford Nanopore, 100X et 70X Illumina, pour n'en nommer que quelques-unes. Les avancées technologiques en matière de séquençage ont conduit au séquençage des 8% restants comme mentionné ci-dessus.
La seule limitation de la séquence T2T-CHM13 est l'absence d'un chromosome Y. Ce séquençage est actuellement en cours, en utilisant l'ADN de la lignée cellulaire HG002, qui possède un 46 (23 paires) avec un caryotype XY. La séquence sera ensuite assemblée en utilisant les mêmes méthodes développées pour le génome homozygote CHM13.
La disponibilité de T2T-CHM13 comme nouvelle référence génome représente une percée majeure qui aidera à comprendre le rôle des régions d'hétérochromatine et à mieux comprendre ses effets sur les processus cellulaires. Jusqu'à ce que le séquençage du chromosome Y soit terminé, celui-ci servira de génome de référence pour de futures études visant à comprendre les processus et les fonctions cellulaires.
***
Bibliographie
- Nurk S, Koren S, Rhie A, Rautiainen M, Bzikadze AV, Mikheenko A et al. La séquence complète d'un génome humain bioRxiv 2021.05.26.445798; EST CE QUE JE: https://doi.org/10.1101/2021.05.26.445798
***